Contents
clear,close all,clc
Cargar valores de entrada y salida del sistema.
load CIP_01_2022.mat
dmin=[0 -1 -1 -1];
dmax=[1 1 1 1];
Ni=length(r);
Ne=2;
Ngen=200*Ni;
Pc=0.9;
Tm=1;
Pm=1;
ts=1.2;
tstcp=10;
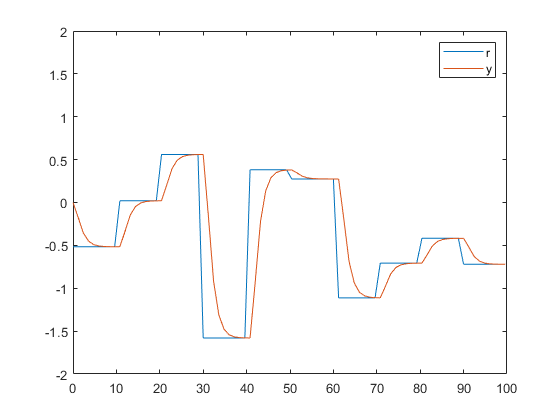
figure;
plot(t,[r y]);
legend('r','y');
ylim([-2 2]);
drawnow;
data.t=t;
data.y=y;
data.r=r;
data.ts=ts;
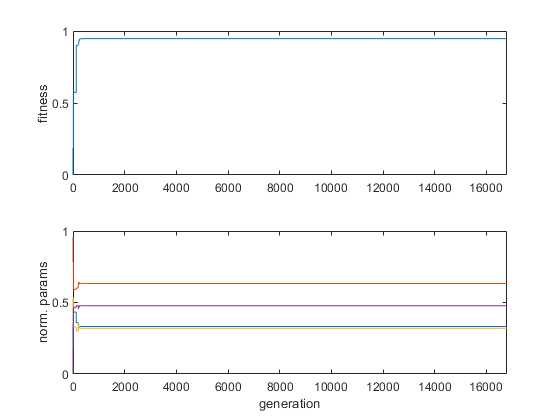
Algoritmo genético asociado a la identificación y ordenamiento
tic
[indop,Fop]=agFDT(@fitness,dmin,dmax,Ni,Ne,Ngen,Pc,Pm,Tm,data);
toc
iop=indop(end,:);
Elapsed time is 53.983220 seconds.
Seleccion del valor de adaptacion optimo
fop=Fop(end)
y;
fop =
0.9467
Resultados de la funcion de transferencia identificada.
a=iop(1);
b=iop(2);
c=iop(3);
d=iop(4);
gzm=tf([a b],[1 c d],ts)
gsm=d2c(gzm,'zoh')
ym=lsim(gzm,r,t);
gzm =
0.332 z + 0.2659
------------------------
z^2 - 0.3601 z - 0.04523
Sample time: 1.2 seconds
Discrete-time transfer function.
Warning: The model order was increased to handle real negative poles.
gsm =
0.1651 s^2 - 1.03 s + 6.909
---------------------------------
s^3 + 4.511 s^2 + 13.09 s + 6.872
Continuous-time transfer function.
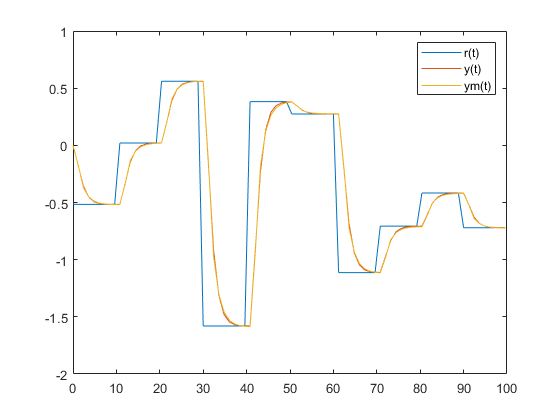
Se muestran los resultados de r, y, ym.
figure;
plot(t,[r y ym]);
legend('r(t)','y(t)','ym(t)');